ژن گزارشگر
در زیستشناسی مولکولی، ژن گزارشگر (غالباً به سادگی گزارشگر) ژنی است که محققان آن را به دنباله تنظیم کننده (سکانس تنظیم کننده) ژن مورد نظر در باکتریها، سلول سلولی، حیوانات یا گیاهان، پیوند میدهند. به این ژنها گزارشگر گفته میشود زیرا خصوصیاتی که به ارگانیسمهای بیان کننده آنها میدهند به راحتی شناسایی و اندازهگیری میشوند یا اینکه نشانگرهای قابل انتخاب هستند. از ژنهای گزارشگر اغلب به عنوان نشانه ای استفاده میشود که آیا ژن خاصی توسط سلول یا ارگانیسم جذب شده آیا در آن بیان شدهاست؟.
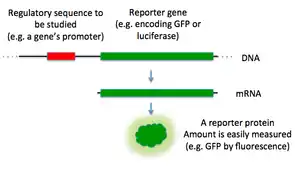
ژنهای گزارشگر رایج
دانشمندان برای تزریق ژن گزارشگر به ارگانیسم، ژن گزارشگر و ژن مورد نظر را در همان سازه DNA (با سازه دبل هلیکس ژنوم اشتباه نشود، سازه دی.ان. آ یک ساختار مهندسی شدهاست که برای اهداف تراریختگی مورد استفاده قرار میگیرد) قرار میدهند تا در سلول یا ارگانیسم قرار گیرد. برای باکتریها یا سلولهای پروکاریوتی در فرهنگ، این معمولاً به شکل یک مولکول DNA حلقوی به نام پلاسمید است. استفاده از ژن گزارشگر که به صورت بومی در سلول یا ارگانیسم مورد مطالعه بیان نشده باشد بسیار مهم است، زیرا بیان خبرنگار به عنوان یک نشانگر برای جذب موفقیتآمیز ژن مورد استفاده است.[1]
ژنهای گزارشگر معمولاً مورد استفاده که ویژگیهای قابل رویت بصری را القا میکنند معمولاً شامل پروتئینهای فلورسنت و لومینسانس هستند. به عنوان مثال میتوان به ژن رمزگذار پروتئین فلورسنت سبز (GFP) چتر دریایی (ماهی ژله ای) اشاره کرد که باعث میشود سلولهایی که آن را بیان میکنند در زیر نور آبی سبز بدرخشند، آنزیم لوسیفراز که با تولید لوسیفرین واکنش را برای تولید نور و پروتئین فلورسنت قرمز از ژن dsRed[2][3][4][5][6] ژن GUS معمولاً در گیاهان مورد استفاده قرار میگیرد اما لوسیفراز و GFP بیشتر رایج هستند.[7][8]
گزارشگر رایج در باکتریها ژن E. coli lacZ است که پروتئین بتا-گالاکتوزیداز را کد میکند.[9] این آنزیم باعث میشود باکتریهایی که ژن را بیان میکنند وقتی روی محیطی که حاوی X-gal آنالوگ سوبسترا است رشد کنند آبی به نظر میرسند. نمونه ای از یک نشانگر قابل انتخاب که گزارشگر در باکتریها نیز میباشد ژن کلرامفنیکل استیل ترانسفراز (CAT) است که مقاومت به آنتیبیوتیک کلرامفنیکل را ایجاد میکند.[10]
| نام ژن | محصول ژن | سنجش | مرجع. |
|---|---|---|---|
| lacZ | β- گالاکتوزیداز | سنجش آنزیم، هیستوشیمیایی | [9] |
| گربه | کلرامفنیکل استیل ترانسفراز | استیلاسیون کلرامفنیکل | [10] |
| gfp | پروتئین فلورسنت سبز | فلورسنت | [2] |
| rfp | پروتئین فلورسنت قرمز | میکروسکوپی ،اسپکتروفتومتری | [11] |
| لوک | آنزیم لوسیفراز | بیولومینسانس | [3] |
سنجشهای تحول و انتقال
بسیاری از روشهای ترانسفکشن (ترا آلودگی) و تحول - دو روش بیان ژن خارجی یا اصلاح شده در ارگانیسم - فقط در درصد کمی از جمعیتی که تحت این تکنیکها قرار دارند کارآمدتر است.[12][13] بنابراین، روشی برای شناسایی آن چند رویداد موفق جذب ژن لازم است. ژنهای گزارشگر مورد استفاده در این روش بهطور معمول تحت پروموتر خود (مناطق DNA ای که رونویسی ژن را آغاز میکنند) مستقل از ژن معرفی شده مورد علاقه بیان میشوند. ژن گزارشگر را میتوان بصورت سازنده بیان کرد (یعنی "همیشه روشن است") یا با یک مداخله خارجی مانند معرفی ایزوپروپیل β-D-1-تیوگالاکتوپیروزانوزید (IPTG) در سیستم β-galactosidase القا میشود.[9] در نتیجه، بیان ژن گزارشگر مستقل از بیان ژن مورد علاقه است، این زمانی یک مزیت است که ژن مورد علاقه فقط تحت شرایط خاص خاص یا در بافتهایی که دسترسی به آنها دشوار است، بیان شود.[1]
در مورد گزارشگرهای نشانگر قابل انتخاب مانند CAT، جمعیت ترانسفکت شده باکتریها را میتوان روی یک بستر که حاوی کلرامفنیکل است، پرورش داد. تنها سلولهایی که سازه حاوی ژن CAT را با موفقیت به دست گرفتهاند در این شرایط زنده میمانند و تکثیر میشوند.[10]
آزمونهای سنجش بیان ژن
از ژنهای گزارشگر میتوان برای سنجش بیان ژن مورد علاقه استفاده کرد که سنجش کمی آن بهطور معمول دشوار است.[1] ژنهای گزارشگر میتوانند پروتئینی تولید کنند که تأثیر واضح یا فوری آن روی محیط کشت سلول یا ارگانیسم باشد. آنها بهطور ایدهآل در ژنوم بومی وجود ندارند تا بتوانند بیان ژن گزارشگر را در نتیجه بیان ژن مورد علاقه جدا کنند.[14]
برای فعال سازی ژنهای گزارشگر، میتوان آنها را به صورت سازنده بیان کرد، جایی که مستقیماً به ژن مورد نظر متصل شده و همجوشی ژن ایجاد میکنند.[15] این روش یک مثال از استفاده از عناصر سیس (cis-acting عناصر توالی نظارتی CIS، توالیهای موجود در منطقه ۳ ′ و ۵ ′ ترجمه نشده، اینترونها یا مناطق کد کننده RNAهای پیش ساز و mRNAهای بالغ هستند که بهطور انتخابی توسط یک مجموعه مکمل از یک یا چند عامل متعامل برای تنظیم ژن پس از رونویسی شناخته میشوند) است که در آن دو ژن تحت اثر یک پروموتر مشابه هستند و رونویسی آنها RNA پیامرسان واحد را بوجود میآورد. mRNA ژن سپش به پروتئین ترجمه میشود. مهم این است که هر دو پروتئین قادر باشند به درستی برابر به رونوشت فعال خود خمش یابند (تا شدگی پروتئین کلمه ای است که در فارسی برای Protein folding پیشنهاد شده و از لحاظ بیولوژی صحیح نمیباشد) و با سوبستراهای خود تعامل کنند. در ساخت سازه DNA، معمولاً بخشی از برنامهنویسی DNA برای یک منطقه پیوند دهنده پلی پپتیدی انعطافپذیر در نظر گرفته میشود تا گزارشگر و محصول ژن فقط اندکی با یکدیگر تداخل داشته باشند.[16][17] ژنهای گزارشگر را میتوان با القا در حین رشد نیز بیان کرد. در این موارد، عناصر ترانس(trans-acting عناصر ترانس عوامل رونویسی یا سایر پروتئینهای متصل به DNA هستند که توالیهای خاص عناصر سیس را برای شروع، تقویت یا سرکوب رونویسی شناسایی و به آنها متصل میشوند) عناصر، مانند عوامل رونویسی استفاده میشود برای بیان ژن گزارشگر.[18][19]
سنجش ژن گزارشگر بهطور فزاینده ای در غربالگری با توان عملیاتی بالا (HTS) برای شناسایی بازدارندههای مولکول کوچک و فعال کنندههای اهداف پروتئین و مسیرهای کشف دارو و شیمی آلی مورد استفاده قرار گرفتهاست. از آنجا که خود آنزیمهای گزارشگر (به عنوان مثال لوسیفراز کرم شب تاب) میتوانند اهداف مستقیم مولکولهای کوچک باشند و تفسیر دادههای HTS را سردرگم کنند، طرحهای جدید گزارشگر تصادفی شامل سرکوب مصنوع ساخته شدهاست.[20][21]
سنجشهای پروموتر
از ژنهای گزارشگر میتوان برای ارزیابی فعالیت یک پروموتر خاص در سلول یا ارگانیسم استفاده کرد.[22] در این حالت «ژن مورد علاقه» جداگانه ای وجود ندارد. ژن گزارشگر به سادگی تحت کنترل پروموتر هدف قرار میگیرد و فعالیت محصول ژن گزارشگر کمی اندازهگیری میشود. نتایج بهطور معمول نسبت به فعالیت تحت یک پروموتر «اجماع» شناخته شده برای القای بیان ژن قوی گزارش میشود.
موارد استفاده بیشتر
استفاده پیچیدهتر از ژنهای گزارشگر در مقیاس بزرگ در غربالگری دو هیبریدی است، که هدف آن شناسایی پروتئینهایی است که بومی با یکدیگر در داخل بدن تعامل دارند.[23]
جستارهای وابسته
- سیستم گزارشگر GUS
منابع
- Debnath, Mousumi; Prasad, Godavarthi B.K.S.; Bisen, Prakash S. (2010), Debnath, Mousumi; Prasad, Godavarthi B.K.S.; Bisen, Prakash S., eds., "Reporter Gene", Molecular Diagnostics: Promises and Possibilities, Springer Netherlands: 71–84, doi:10.1007/978-90-481-3261-4_5, ISBN 978-90-481-3261-4
- Soboleski, Mark R.; Oaks, Jason; Halford, William P. (March 2005). "Green fluorescent protein is a quantitative reporter of gene expression in individual eukaryotic cells". The FASEB Journal. 19 (3): 440–442. doi:10.1096/fj.04-3180fje. ISSN 0892-6638. PMC 1242169. PMID 15640280.
- Smale, S. T. (2010-05-01). "Luciferase Assay". Cold Spring Harbor Protocols. 2010 (5): pdb.prot5421. doi:10.1101/pdb.prot5421. ISSN 1559-6095. PMID 20439408.
- Jach, Guido; Binot, Elke; Frings, Sabine; Luxa, Kerstin; Schell, Jeff (2001). "Use of red fluorescent protein from Discosoma sp. (dsRED) as a reporter for plant gene expression". The Plant Journal. 28 (4): 483–491. doi:10.1046/j.1365-313X.2001.01153.x. ISSN 1365-313X. PMID 11737785.
- Zhang, Qixiang; Walawage, Sriema L.; Tricoli, David M.; Dandekar, Abhaya M.; Leslie, Charles A. (May 2015). "A red fluorescent protein (DsRED) from Discosoma sp. as a reporter for gene expression in walnut somatic embryos". Plant Cell Reports. 34 (5): 861–869. doi:10.1007/s00299-015-1749-1. ISSN 1432-203X. PMID 25627255.
- Mikkelsen, Lisbeth; Sarrocco, Sabrina; Lübeck, Mette; Jensen, Dan Funck (2003-06-01). "Expression of the red fluorescent protein DsRed-Express in filamentous ascomycete fungi". FEMS Microbiology Letters. 223 (1): 135–139. doi:10.1016/S0378-1097(03)00355-0. ISSN 0378-1097. PMID 12799012.
- Hull, Gillian A.; Devic, Martine (1995), Jones, Heddwyn, ed., "The β-Glucuronidase (gus) Reporter Gene System", Plant Gene Transfer and Expression Protocols, Methods in Molecular Biology™, Springer New York, 49: 125–141, doi:10.1385/0-89603-321-x:125, ISBN 978-1-59259-536-5, PMID 8563799
- Koo, J.; Kim, Y.; Kim, J.; Yeom, M.; Lee, I. C.; Nam, H. G. (2007). "A GUS/Luciferase Fusion Reporter for Plant Gene Trapping and for Assay of Promoter Activity with Luciferin-Dependent Control of the Reporter Protein Stability". Plant and Cell Physiology. 48 (8): 1121–1131. doi:10.1093/pcp/pcm081. PMID 17597079.
- Smale, S. T. (2010-05-01). "-Galactosidase Assay". Cold Spring Harbor Protocols. 2010 (5): pdb.prot5423. doi:10.1101/pdb.prot5423. ISSN 1559-6095. PMID 20439410.
- Smale, S. T. (2010-05-01). "Chloramphenicol Acetyltransferase Assay". Cold Spring Harbor Protocols. 2010 (5): pdb.prot5422. doi:10.1101/pdb.prot5422. ISSN 1559-6095. PMID 20439409.
- Nordgren, I. K.; Tavassoli, A (2014). "A bidirectional fluorescent two-hybrid system for monitoring protein-protein interactions". Molecular BioSystems. 10 (3): 485–90. doi:10.1039/c3mb70438f. PMID 24382456.
- Hanahan, Douglas; Jessee, Joel; Bloom, Fredric R. (1991-01-01), "[4] Plasmid transformation of Escherichia coli and other bacteria", Methods in Enzymology, Bacterial Genetic Systems, Academic Press, 204: 63–113, doi:10.1016/0076-6879(91)04006-a, ISBN 978-0-12-182105-0, PMID 1943786
- Hanahan, Douglas (1983-06-05). "Studies on transformation of Escherichia coli with plasmids". Journal of Molecular Biology. 166 (4): 557–580. doi:10.1016/S0022-2836(83)80284-8. ISSN 0022-2836. PMID 6345791.
- Promega Corporation, Promega Corporation (October 22, 2014). "Introduction to Reporter Gene Assays". YouTube. Retrieved March 21, 2020.
- de Jong, Hidde; Geiselmann, Johannes (2015). Maler, Oded; Halász, Ádám; Dang, Thao; Piazza, Carla, eds. "Fluorescent Reporter Genes and the Analysis of Bacterial Regulatory Networks". Hybrid Systems Biology. Lecture Notes in Computer Science. Springer International Publishing. 7699: 27–50. doi:10.1007/978-3-319-27656-4_2. ISBN 978-3-319-27656-4.
- Spector, David L.; Goldman, Robert D. (2006-12-01). "Constructing and Expressing GFP Fusion Proteins". CSH Protocols. 2006 (7): pdb.prot4649. doi:10.1101/pdb.prot4649. PMID 22484672.
- Chen, Xiaoying; Zaro, Jennica; Shen, Wei-Chiang (2013-10-15). "Fusion Protein Linkers: Property, Design and Functionality". Advanced Drug Delivery Reviews. 65 (10): 1357–1369. doi:10.1016/j.addr.2012.09.039. ISSN 0169-409X. PMC 3726540. PMID 23026637.
- Hanko, Erik K. R.; Minton, Nigel P.; Malys, Naglis (2019-01-01), Shukla, Arun K., ed., "Chapter Nine - Design, cloning and characterization of transcription factor-based inducible gene expression systems", Methods in Enzymology, Chemical and Synthetic Biology Approaches To Understand Cellular Functions - Part A, Academic Press, 621: 153–169, doi:10.1016/bs.mie.2019.02.018, PMID 31128776, retrieved 2019-12-16
- Kallunki, Tuula; Barisic, Marin; Jäättelä, Marja; Liu, Bin (2019-07-30). "How to Choose the Right Inducible Gene Expression System for Mammalian Studies?". Cells. 8 (8): 796. doi:10.3390/cells8080796. ISSN 2073-4409. PMC 6721553 Check
|pmc=value (help). PMID 31366153. - Cheng, K.C.; Inglese, J. (2012). "A coincidence reporter-gene system for high throughput screening". Nature Methods. 9 (10): 937. doi:10.1038/nmeth.2170. PMC 4970863. PMID 23018994.
- Hasson, S.A.; Fogel, A.I.; Wang, C.; MacArthur, R.; Guha, R.; Heman-Ackahc, S.; Martin, S.; Youle, R.J.; Inglese, J. (2015). "Chemogenomic profiling of endogenous PARK2 expression using a genome-edited coincidence reporter". ACS Chem. Biol. 10 (5): 1188–1197. doi:10.1021/cb5010417. PMID 25689131.
- Jugder, Bat-Erdene; Welch, Jeffrey; Braidy, Nady; Marquis, Christopher P. (2016-07-26). "Construction and use of aCupriavidus necatorH16 soluble hydrogenase promoter (PSH) fusion togfp(green fluorescent protein)". PeerJ. 4: e2269. doi:10.7717/peerj.2269. ISSN 2167-8359. PMC 4974937. PMID 27547572.
- Brückner, Anna; Polge, Cécile; Lentze, Nicolas; Auerbach, Daniel; Schlattner, Uwe (2009-06-18). "Yeast Two-Hybrid, a Powerful Tool for Systems Biology". International Journal of Molecular Sciences. 10 (6): 2763–2788. doi:10.3390/ijms10062763. ISSN 1422-0067. PMC 2705515. PMID 19582228.