تشخیص به کمک کامپیوتر
شناسایی با کمک کامپیوتر (Computer-aided detection: CADe)، که به آن تشخیص با کمک کامپیوتر (Computer-aided diagnosis: CADx) نیز گفته میشود، سیستمهایی هستند که در تفسیر تصاویر پزشکی، به پزشکان یاری میرسانند. تکنیکهای پرتو ایکس, MRI، و سیستمهای فراصوت، اطلاعات زیادی ایجاد میکنند که رادیولوژیست یا پزشک حرفهای باید آنها را در زمان کوتاهی تحلیل و ارزیابی کند. سیستمهای CAD، تصاویر دیجیتال را به منظور تسهیل تصمیمگیری برای پزشک، پردازش کرده و بخشهای مشکوک آن، مانند بیماریهای احتمالی، را مشخص میکنند.
با تشخیص (هوش مصنوعی) اشتباه گرفته نشود.
| تشخیص به کمک کامپیوتر | |
|---|---|
| تشخیص پزشکی | |
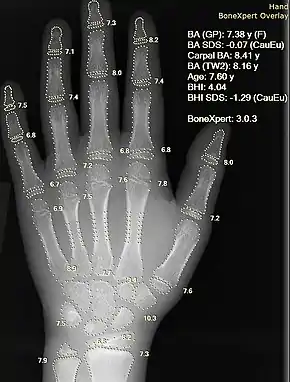 رادیوگرافی دست با استفاده از محاسبه خودکار عصر استخوان توسط نرمافزار کامپیوتری | |
| دلیل | تشخیص تصویرها به کمک کامپیوتر |
علاوه بر این، با ظهور تصویربرداری تمام-لغزشی (whole-slide imaging) و الگوریتمهای یادگیری ماشین (machine learning algorithms) میتوان در آینده از CAD در آسیبشناسی دیجیتال (digital pathology) بهره برد. تاکنون کاربرد آن تنها به سنجش کیفیت ایمنسازی (immunostaining) محدود شده، اما از آن میتوان در استاندارد H&S stain نیز استفاده کرد.[1]
CAD، یک تکنولوژی میانرشته بوده که هوش مصنوعی و بینایی ماشین را با پردازش تصاویر رادیولوژی و آسیبشناسی (pathology) ترکیب میکند. یکی از کاربردهای معمول آن، شناسایی تومور است. بهطور مثال، در برخی بیمارستانها از CAD برای معاینههای پزشکی پیشگیرانه در ماموگرافی (تشخیص سرطان پستان)، تشخیص پلیپ روده بزرگ و سرطان ریه استفاده میکنند.
معمولاً سیستمهای شناسایی کامپیوتری (CADe) به نشانه گذاری ساختارها و بخشهای مشکوک منحصر میشوند. سیستمهای تشخیص کامپیوتری (CADx)، ساختارهای مشکوک را ارزیابی میکنند. برای مثال، در ماموگرافی، CAD، خوشههای microcalcification و ساختارهای پرچگال (hyperdense) را در بافت نرم علامت گذاری میکند که به رادیولوژیست امکان نتیجهگیری در مورد شرایط آسیبشناسی را میدهد. یکی دیگر از کاربردها، CADq است که اطلاعاتی همچون اندازه تومور یا رفتار تومور در برابر جذب متوسط را کمی میکند. ردهبندی درمانی ساده با استفاده از کامپیوتر (Computer-aided simple triage: CAST) یکی دیگر از انواع CAD است که عمل تفسیر اولیه تمام خودکار و ردهبندی مطالعات به دستههای معنی دار (برای مثال مثبت و منفی) را انجام میدهد. از CAST تنها میتوان در تصاویر تشخیصی اورژانسی، که به تشخیص فوری شرایط بحرانی و تهدیدکننده زندگی نیاز داریم، استفاده کرد.
بیش از ۴۰ سال است که از CAD در محیطهای پزشکی استفاده میشود؛ با این حال نمیتوان آن را جایگزین پزشک یا متخصصان دیگر دانست و تنها نقش پشتیبانی را ایفا میکند. بهطور کلی، متخصص (معمولاً یک رادیولوژیست) مسئول تفسیر نهایی تصویر پزشکی است. هدف بعضی از سیستمهای CAD، شناسایی اولین نشانههای غیرطبیعی در انسان است که متخصص قادر به تشخیص آنها نمیباشد؛ مانند رتینوپاتی دیابتی، تحریف ساختاری در ماموگرام،[2][3] گرههای شیشه ای در تصاویر CT قفسه سینه[4][5] و ضایعات غیر پلیپی (مسطح) در کلونوگرافی CT.[6]
موضوعات
روششناسی
CAD اساساً از بازشناسی الگوی به شدت پیچیده استفاده میکند. تصاویر پرتو ایکس و سایر تصاویر برای یافتن ساختارهای مشکوک اسکن میشوند. بهطور معمول برای بهینه کردن الگوریتم، چندین هزار تصویر مورد نیاز است. دادههای تصاویر دیجیتال با فرمت دایکام در سرور CAD کپی شده و طی چندین مرحله، آمادهسازی و تحلیل میشوند.
۱. پیش پردازش:
- کاهش درست نماها (اشکالات در تصاویر)
- کاهش نویز تصویر
- سطحبندی (هماهنگ سازی) کیفیت تصاویر (بهبود کنتراست) برای از بین بردن شرایط پایه ای مختلف تصویر مانند تفاوت پارامترهای پرتودهی
- فیلتر کردن
۲. تقطیع (segmentation):
- شناسایی ساختارهای مختلف در تصویر مانند قلب، ریه، قفسه سینه، رگهای خونی و ضایعات احتمالی.
- تطبیق با بانک اطلاعات آناتومیک
- نمونه برداری از سطوح خاکستری در حجم دلخواه[7]
۳. تحلیل ساختار/ ROI (ناحیه دلخواه): ویژگیهای خاص هر یک از نواحی شناخته شده بهطور جداگانه تحلیل میشود که این ویژگیها از قرار زیر است:
- میزان فشرده بودن
- شکل، اندازه و محل قرارگیری
- مراجعه به ساختارها/ROIهای نزدیک
- بررسی میانگین سطوح خاکستری داخل یک ROI
- متناسب کردن سطوح خاکستری با مرز ساختارهای داخل ROI
۴. ارزیابی/ طبقهبندی: پس از اینکه ساختار مورد تحلیل قرار گرفت، هر یک از ROIها به صورت جداگانه برای بررسی احتمال TP، ارزیابی میشوند. در ادامه نمونههایی از الگوریتمهای طبقهبندی ذکر خواهد شد:
- قوانین نزدیکترین همسایه (به عنوان مثال، kامین همسایه نزدیک)[8]
- طبقهبندی حداقل فاصله (Minimum distance classifier)
- طبقهبندی آبشاری
- طبقهبندی بیزی ساده
- شبکه عصبی مصنوعی[9][10]
- شبکه تابع پایه شعاعی (RBF)
- ماشین بردار پشتیبانی (SVM)[11][12]
- تجزیه و تحلیل اجزای اصلی (PCA)
در صورتی که ساختارهای شناسایی شده به حد آستانه مشخصی برسند، در تصویر علامت گذاری خواهند شد. این علامت گذاریها، بسته به نوع سیستم CAD، بهطور دائم یا موقت است. مزیت بعدی این است که تنها علامت گذاریهای تاییدشده توسط رادیولوژیست ذخیره خواهد شد. علامت گذاریهای غلط نباید ذخیره شود، زیرا معاینات بعدی را دشوار میکند.
حساسیت و ویژگی
سیستمهای CAD، به دنبال علامت گذاری ساختارهای مشکوک هستند. در حال حاضر این سیستمها قادر به شناسایی ۱۰۰٪ تغییرات پاتولوژیک نیستند. نرخ ضربه (حساسیت)، بسته به نوع سیستم و کاربرد آن میتواند بیش از ۹۰٪ باشد.[13] به تشخیصهای درست، مثبت واقعی (True Positive: TP) و به علامت گذاری نادرست بخشهای سالم، مثبت کاذب (False Positive: FP) گفته میشود. هرچه میزان FPها کمتر باشد، نشان دهنده بالاتر بودن ویژگی (specificity) است. کم بودن ویژگی، مقبولیت سیستم CAD را کاهش میدهد، زیرا کاربر باید همه این تشخیصهای نادرست را پیدا کند. در آزمایشهایی که برای بررسی کلی ریه هستند (CAD قفسه سینه)، میتوان نرخ FP را به ۲ مورد در هر آزمایش کاهش داد. در سایر بخشها (مثلاً آزمایشهای CT ریه)، نرخ FP میتواند ۲۵ یا بیشتر باشد. در سیستمهای CAST، به منظور ردهبندی معنی دار مطالعات، نرخ FP باید بسیار کم باشد (کمتر از ۱ مورد در هر آزمایش).
نرخ شناسایی مطلق
نرخ شناسایی مطلق رادیولوژیست، معیاری جایگزین برای حساسیت و ویژگی است. بهطور کلی، نتایج آزمایشهای پزشکی در مورد حساسیت، ویژگی و نرخ شناسایی مطلق، به شدت متغیر میباشد. نتایج هر مطالعه وابسته به شرایط پایه ای آن بوده و باید بر اساس آن شرایط ارزیابی شود. مواد ذکر شده در ادامه، تأثیر زیادی دارد:
- طراحی گذشته نگر (Retrospective) یا آینده نگر (prospective)
- کیفیت تصاویر مورد استفاده
- شرایط آزمایش اشعه ایکس
- تجربه و آموزش رادیولوژیست
- نوع ضایعه
- اندازه ضایعه مورد نظر
کاربردها

از سیستم CAD میتوان در تشخیص سرطان پستان، سرطان ریه، سرطان روده، سرطان پروستات، متاستاز استخوان، بیماری سرخرگ کرونری، نقص مادرزادی قلب، آسیبشناسی مغز، آلزایمر و رتینوپاتی دیابتی، استفاده کرد.
سرطان پستان
از CAD در غربالگری ماموگرافی (آزمایش x-ray سینه زنان) استفاده میشود. غربالگری ماموگرافی برای تشخیص زودهنگام سرطان پستان مورد استفاده قرار میگیرد. سیستم CAD به طبقهبندی تومورهای خوشخیم و بدخیم کمک میکند. این سیستم بیشتر در آمریکا و هلند به کار گرفته شدهاست. اولین سیستم CAD برای ماموگرافی، طی یک پروژه تحقیقاتی در دانشگاه شیکاگو ایجاد شد. امروزه، این سیستم توسط iCAD و Hologic ارائه میشود. سیستمهای CAD، علیرغم برخورداری از حساسیت بالا، ویژگی (specificity) بسیار کمی دارند؛ این امر موجب نامشخص بودن مزایای استفاده از CAD میشود. در سال ۲۰۰۸، یک بررسی اصولی بر روی غربالگری ماموگرافی با استفاده از CAD به این نتیجه دست یافت که CAD نمیتواند اثر معنی داری بر نرخ تشخیص سرطان داشته باشد، اما نرخ recall (یعنی نرخ FP) را به صورت ناخواسته افزایش میدهد؛ با این حال، در مطالعات مختلف، ناهمگونی قابل توجهی را در تأثیر بر recall نشان داد.[14]
علاوه بر این، روشهایی برای ارزیابی ماموگرافی بر اساس تصویربرداری رزونانس مغناطیسی نیز در دسترس است.
سرطان ریه
توموگرافی کامپیوتری (computed tomography) به همراه سیستم CAD سه بعدی، به عنوان روشی ثانویه در تشخیص سرطان ریه در نظر گرفته میشود.[15] در این مورد، پایگاه دادهای با ۳۰۰۰ تصویر فراهم و بررسی شدهاست. با این روش، ضایعات گرد (سرطان ریه، متاستاز و تغییرات خوشخیم) با اندازه بزرگتر از ۱ میلیمتر قابل شناسایی است. امروزه همه فروشندگان معروف سیستمهای پزشکی این سیستم را پیشنهاد میکنند.
تشخیص زودهنگام سرطان ریه بسیار ارزشمند است. با این وجود تشخیص سرطان ریه در مراحل ابتدایی (مرحله ۱) بسیار دشوار است. ضایعات گرد با اندازه ۵–۱۰ میلیمتر به آسانی نادیده گرفته میشوند.[16] به کار بردن سیستمهای CAD قفسه سینه، میتواند به شناسایی تغییرات کوچک کمک کند. تعدادی از محققان، سیستم CAD را برای شناسایی گرههای ریه (ضایعات گرد کوچکتر از ۳۰ میلیمتر) در رادیوگرافی قفسه سینه[17][18][19] و CT,[20][21] و سیستمهای CAD تشخیصی گرههای ریه (مثلاً تمایز بین خوشخیم و بدخیم) در CT، توسعه دادند. تصویربرداری مجازی انرژی دوگانه (Virtual dual-energy imaging)[22][23][24][25] عملکرد سیستمهای CAD در رادیوگرافی قفسه سینه را بهبود بخشیدهاست.[26]
سرطان روده
CAD برای تشخیص پولیپهای کولورکتال در کولون در کلونوگرافی سی تی موجود است.[27][28] پلیپها رشد کمی داشته و از پوشش داخلی روده به وجود میآیند. CAD، پلیپها را از روی شکل برآمده آنها تشخیص میدهد. برای جلوگیری از رخ دادن مثبت کاذب، CAD دیواره روده و چینهای haustral را نادیده میگیرد.
بیماری عروق کرونر
CAD برای شناسایی خودکار بیماری های شریان کروناری در CT آنژیوگرافی کروناری (coronary CT angiography: CCTA) در دسترس است.
نقص مادرزادی قلب
شناسایی زودهنگام بیماریها میتواند مرز بین مرگ و زندگی باشد. CADe را میتوان از طریق گوش دادن با یک گوشی پزشکی دیجیتال (digital stethoscope) یا نرمافزارهای تخصصی انجام داد، که به آن شنود درمانی با کمک کامپیوتر (Computer-aided auscultation) نیز میگویند. با استفاده از این روش، مرمرها (Murmurs) یا صداهای غیرطبیعی قلب، که به علت جریان خون در قسمت معیوب قلب ایجاد میشوند را میتوان با حساسیت و ویژگی بالایی شناسایی کرد. شنود درمانی با کمک کامپیوتر به نویز خارجی و صداهای بدن حساس بوده و در نتیجه برای صحت عملکرد به محیطی ساکت نیازمند است.
شناسایی آسیب های مغز
چپلوت، همکارانش اولین کسانی بودند که برای شناسایی آسیبهای مغز از ضرایب تبدیل موجک گسسته (DWT) استفاده کردند.[29] مایترا (Maitra) و چترجی (Chatterjee)، تبدیل اسلنتلت (Slantlet transform) را در این مورد به کار بردند که نسخه بهبود یافته DWT است. آنها بردار ویژگی هر تصویر را، با در نظر گرفتن دامنه خروجی تبدیل اسلنتلت مربوط به شش موقعیت مکانی که مطابق منطقی خاص انتخاب شده بود، محاسبه کردند.[30]
در سال ۲۰۱۰، ونگ (Wang) و وو (Wu) روشی مبتنی بر شبکه عصبی رو به جلو (forward neural network: FNN)، جهت تقسیمبندی تصاویر MR مغز به دو گروه عادی و غیرعادی، ارائه کردند. پارامترهای FNN توسط روش بهینهسازی خودکار گروه ذرات بینظم (adaptive chaotic particle swarm optimization: ACPSO) بهینه شد. نتایج به دست آمده بر روی ۱۶۰ تصویر نشان داد که صحت این طبقهبندی ۹۸٫۷۵٪ بود.[31]
در سال ۲۰۱۱، وو و ونگ روشی ارائه کردند که در آن از DWT برای استخراج ویژگی، از PCA برای کاهش بعد ویژگی و از FNN به همراه روش لانه زنبوری بی نظم ساختگی درجهبندی شده (scaled chaotic artificial bee colony: SCABC) به عنوان طبقهبند استفاده کردند.[32]
در سال ۲۰۱۳، ساریتا (Saritha) و همکارانش نخستین افرادی بودند که برای شناسایی آسیبهای مغز، آنتروپی ویولت (wavelet entropy: WE) را به کار بردند. علاوه بر این، ساریتا پیشنهاد کرد که از نمودارهای شبکه عنکبوتی استفاده کنند.[33] پس از آن، ژانگ (Zhang) و همکارانش، ثابت کردند که حذف نمودارهای شبکه عنکبوتی، تأثیری بر عملکرد ندارد.[34] روش جست جوی الگوهای ژنتیک، برای شناسایی مغز آسیب دیده از گروه کنترل، اعمال شد. صحت طبقهبندی این روش، ۹۵٫۱۸۸٪ گزارش شد.[35] داس (Das) و همکارانش، از تبدیل ریپلت (Ripplet transform) استفاده کردند.[36] ژانگ و همکارانش پیشنهاد استفاده از روش بهینهسازی گروه ذرات (paricle swarm optimization: PSO) را دادند.[37] کلبخانی (Kalbkhani) و همکارانش استفاده از مدل GARCH را پیشنهاد کردند.[38]
در سال ۲۰۱۴، الدحشان (El-Dahshan) و همکارانش از شبکه عصبی دو پالسی (pulse coupled neural network) استفاده کردند.[39]
در سال ۲۰۱۵، ژو (Zhou) و همکارانش پیشنهاد دادند که طبقهبند Naïve Bayesian برای شناسایی آسیبهای مغز استفاده شود.[40]
بیماری آلزایمر
از سیستمهای CAD میتوان برای شناسایی موردهای آلزایمری یا مبتلا به اختلالات شناختی خفیف استفاده کرد.
در سال ۲۰۱۴، پادما (Padma) و همکارانش از ترکیب ویژگیهای بافت آماری ویولت برای تقطیع و طبقهبندی تومورهای AD خوشخیم و بدخیم استفاده کردند.[41] ژانگ و همکارانش دریافتند که صحت طبقهبند درخت تصمیمگیری ۸۰٪، با میانگین زمان محاسبه ۰٫۰۲۲ ثانیه برای هر طبقهبندی است.[42]
Eigenbrain، یک ویژگی جدید مغز برای کمک به شناسایی AD، بر اساس تحلیل اجزای اصلی (Principal Component Analysis)[43] یا تحلیل اجزای مستقل (Independent Component Analysis) است.[44] ثابت شدهاست که SVM چندجمله ای، صحت خوبی دارد. KSVM چندجمله ای بهتر از SVM خطی و SVM کرنل RBF عمل میکند.[45] روشهای دیگر با نتایج قابل قبول شامل استفاده از تحلیل بافت (texture analysis),[46] ویژگیهای ریخت شناختی (morphological features),[47] یا ویژگیهای آماری درجه بالا میشود.[48]
پزشکی هسته ای
سیستم CADx برای تصاویر پزشکی هسته ای در دسترس است. سیستمها تجاری CADx برای تشخیص متاستاز استخوان در اسکنهای کل بدن و بیماریهای شریان کروناری در تصاویر تزریق وریدی قلب وجود دارند.[49]
سیستمهای شناسایی خودکار ضایعات با کمک کامپیوتر، با دارا بودن حساسیت بالا و نرخ قابل قبولی از شناسایی غلط ضایعات، مفید شناخته شده و احتمالاً در آینده برای کمک به تشخیص ضایعات استخوانی در پزشکی هسته ای به کار گرفته خواهدشد.[50]
رتینوپاتی دیابتی
رتینوپاتی دیابتی، یکی از بیماریهای شبکیه است که عمدتاً توسط تصاویر فوندوسکوپی تشخیص داده میشود. بیماران دیابتی در کشورهای صنعتی معمولاً در این شرایط تحت غربالگری منظم قرار میگیرند. برای تشخیص زودهنگام رگهای خونی غیرعادی شبکیه، از تصویر برداری استفاده میشود. تحلیل دستی این تصاویر، وقت گیر و غیرقابل اطمینان است.[51][52] به منظور بالا بردن صحت، حساسیت و ویژگی روش تشخیص خودکار از CAD استفاده میشود. جایگزین کردن نیروی انسانی با سیستمهای CAD میتواند ایمن و مقرون به صرفه باشد.[52]
مراحل اصلی الگوریتم CAD شامل پیش پردازش، استخراج ویژگی و طبقهبندی است.[53]
روشهای پیش پردازش
نرمالیزه کردن تصاویر، تغییرات را در کل تصویر به حداقل میرساند. تغییرات شدت در نواحی بین کنارهها تا مرکز شبکیه چشم موجب عدم صحت تقطیع رگها میشود.[54] بر اساس مطالعه مروری که در سال ۲۰۱۴ انجام شده، در اکثر مطالعات (۱۱ مطالعه از بین ۴۰ مورد تحقیق انجام شده از سال ۲۰۱۱) از این روش استفاده کردهاند.[53]

همسان سازی هیستوگرام، روشی برای افزایش کنتراست تصویر است.[56] از این تکنیک برای افزایش کنتراست محلی استفاده میشود. پس از پایان پردازش، نواحی تاریک در تصویر ورودی، روشنتر خواهد شد که این امر ویژگیهای موجود در آن ناحیه را بهبود میبخشد. از سوی دیگر، نواحی روشنتر در تصویر ورودی، روشن باقیمانده یا از روشنایی آنها کاسته میشود تا با نواحی دیگر تصویر همسان شود. در کنار تقطیع رگها، ویژگیهای دیگر رتینوپاتی دیابتی را میتوان با این تکنیک پیش پردازش جداسازی کرد. میکروآنوریسمها و لختههای خونی، ضایعاتی قرمز رنگ هستند در حالی که ترشحات، نقاط زرد رنگ میباشند. افزایش کنتراست بین این دو گروه، دیدن ضایعات را راحتتر میکند. مطالعه مروری در سال ۲۰۱۴ نشان داد که ۱۰ مورد از ۱۴ مطالعه اخیر (از سال ۲۰۱۱) از این روش استفاده کردهاند.[53]
فیلتر کانال سبز، تکنیک مفید دیگری برای تمایز ضایعات از رگها بوده و به دلیل ایجاد حداکثر کنتراست بین ضایعات مرتبط با رتینوپاتی دیابتی بسیار مهم است.[57] میکروآنوریسمها و لختههای خونی ضایعاتی قرمز رنگ هستند که پس از اعمال فیلتر کانال سبز تیره خواهند شد. در مقابل، ترشحات، نقاط زرد رنگی بوده که پس از فیلتر کردن به صورت نقاط روشن دیده میشوند. بر اساس مطالعه مروری سال ۲۰۱۴، از این روش در ۲۷ مورد از ۴۰ مقاله منتشر شده در سه سال اخیر، استفاده شدهاست.[53] علاوه بر این، از ترکیب فیلتر کانال سبز و سیستم پنجره گذاری دوگانه (double-windowing system) میتوان در شناسایی نقطه مرکزی دیسک اپتیک (محل خروج اعصاب از چشم) بهره جست.[56]
تصحیح روشنایی غیر یکنواخت، تکنیکی برای تنظیم روشنایی غیر یکنواخت در تصاویر فوندوسکوپی میباشد. ممکن است روشنایی غیر یکنواخت، به علت تغییر ویژگیهای آماری تصویر باشد.[53] این تغییرات، پردازشهای بعدی مانند استخراج ویژگی را تحت تأثیر قرار داده و مهمتر از همه اینکه توسط انسان قابل مشاهده نیست. میتوان از طریق تغییر شدت پیکسل با استفاده از شدت پیکسل اصلی (f)، متوسطگیری محلی(λ) و پیکسلهای مورد نظر (μ)، تصحیح روشنایی غیر یکنواخت (f') را انجام داد (با استفاده از رابطه زیر).[58] سپس با اعمال تبدیل والتر-کلین (Walter-Klein transformation)، روشنایی یکنواخت میشود.[58] این روش در بین روشهای موجود در مقالات مطالعه مروری سال ۲۰۱۴، کمترین کاربرد را دارد.
f' = f + μ – λ
عملیات ریختشناسی، دومین روشی است که در مطالعه مروری سال ۲۰۱۴، کمترین کاربرد را دارد.[53] هدف اصلی این روش، بهبود کنتراست، خصوصاً در نواحی تیرهتر نسبت به پس زمینه است.
استخراج ویژگی و طبقهبندی
پس از انجام پیش پردازش، تصویر با روشهای مختلف محاسباتی تحلیل میشود. با این حال بررسی مقالات نشان میدهد که برخی روشها شامل شامل SVM، چند مقیاس (multi-scale)، دنبال کردن رگ (vessel tracking)، رویکرد رشد ناحیه (region growing approach) و رویکردهای مبتنی بر مدل، برای تقطیع رگ بیشتر مورد استفاده قرار میگیرند.

ماشین بردار پشتیبان، طبقهبندی است که در بیش از ۹۰٪ موارد برای تقطیع استفاده میشود.[59] SVM، یک مدل یادگیری تحت نظارت از تکنیکهای بازشناسی الگو است. الگوریتم آن از طریق ایجاد بزرگترین شکاف بین نمونههای داده عمل میکند. هدف، ایجاد بزرگترین شکاف بین این اجزا، به منظور حداقل کردن خطای طبقهبند است.[60] الگوریتم SVM، جهت جدا کردن اطلاعات مربوط به رگهای خونی، بردارهای پشتیبانی ایجاد میکند که در محیطی نظارت شده، پیکسلهای رگها را از سایر قسمتهای تصویر تفکیک میکند. شناسایی رگهای خونی در تصاویر جدید، به روش مشابه و با استفاده از بردارهای پشتیبان انجام میشود. ترکیب این روش با سایر تکنیکهای پیش پردازش، مانند فیلتر کانال سبز، صحت تشخیص رگهای خونی غیرطبیعی را بهبود میبخشد.[53] برخی از مزایای SVM عبارتند از:[60]
- انعطافپذیری - از لحاظ عملکرد بسیار انعطافپذیر است.
- سادگی - ساده، به ویژه با مجموعه دادههای بزرگ (برای تفکیک داده تنها بردارهای پشتیبان مورد نیاز است.)
رویکرد چند مقیاسی (Multi-scale approach)، رویکردی با چند رزولوشن برای تقطیع رگ است. در رزولوشنهای پایین، میتوان ابتدا رگهای با قطر زیاد را استخراج کرد. با افزایش رزولوشن، شاخههای کوچک تر رگهای قطور را میتوان به سادگی شناسایی کرد؛ بنابراین، یکی از مزایای استفاده از این روش، افزایش سرعت تحلیل است.[51] علاوه بر این، این روش را میتوان برای تصاویر سه بعدی نیز به کار برد. نماینده سطح، یک سطح عادی از رگ است که شناسایی وضعیت غیرعادی سطح رگ را امکانپذیر میکند.[56]
دنبال کردن رگ (vessel tracking)، توانایی الگوریتم برای شناسایی خط مرکزی رگها است. این خطوط مرکزی، حداکثر پیک منحنی رگ است. مرکز رگ را میتوان با استفاده از اطلاعات جهتی فراهم شده از فیلتر گوسی، یافت.[56] رویکردهای مشابه که از مفهوم خط مرکزی استفاده میکنند عبارتند از رویکرد مبتنی بر اسکلت (skeleton-based) و رویکرد تفاضلی مبتنی بر هندسه (differential geometry-based).[51]
رویکرد رشد ناحیه (Region growing approach)، روشی برای شناسایی پیکسلهای همسایه با استفاده از شباهتها است. برای شروع نیاز به یک نقطه مرکزی داریم. دو جز اصلی این روش عبارتند از: شباهت و نزدیکی مکانی. هر یک از پیکسلهای همسایه نقطه مرکزی، در صورت تشابه روشنایی، همنوع با نقطه مرکزی در نظر گرفته شده و به ناحیه افزوده میشود. یکی از معایب این روش، نیاز به انتخاب دستی نقطه مرکزی است که باعث ایجاد ناپایداری در آن میشود.[51] کاربرد این تکنیک در شناسایی دیسک اپتیک نیز میباشد.[56]
رویکردهای مبتنی بر مدل (Model-based approaches)، نماینده را برای استخراج رگها از تصویر به کار میبرند. سه دسته اصلی روشهای مبتنی بر مدل عبارتند از تغییرشکل پذیر (deformable)، پارامتریک و تطبیق الگو (template matching).[51] روشهای تغییرشکل پذیر یک شی را تغییر شکل داده تا با مرزهای شکل داخل تصویر مطابقت یابد. روش پارامتریک از شکلهای هندسی رگ خونی مانند لوله، سیلندر، یا بیضی استفاده میکند. علاوه بر این، ترکیب روش کانتور ماری کلاسیک (Classical snake contour) و اطلاعات توپولوژی رگ خونی نیز یک رویکرد مبتنی بر مدل محسوب میشود.[61] در نهایت، تطبیق الگو، استفاده از یک قالب است، که توسط فرایند تغییر شکل تصادفی با استفاده از حالت مخفی مارکف ۱ مطابقت دارد.
تأثیرات در اشتغال زایی
خودکارشدن تشخیصی پزشکی (به عنوان مثال، اندازهگیری گلبولهای قرمز) سابقه تاریخی دارد.[62] انقلاب یادگیری عمیق (deep learning revolution) در سال ۲۰۱۰، تاکنون AIهای ایجاد کرده که در بسیاری از موارد دقیق تر از رادیولوژیستها و متخصصین پوست هستند و انتظار میرود این شکاف رشد کند. برخی از متخصصین، همچون پزشکها، اثرات این AIها را در رشته پزشکی رد میکنند. در مقابل، بسیاری از اقتصاددانان و کارشناسان هوش مصنوعی اعتقاد دارند که رشتههایی مانند رادیولوژی از بین خواهد رفت و شمار زیادی از رادیولوژیستها بیکار شده یا دستمزد آنها کاهش خواهد یافت؛ در نهایت بیمارستانها رادیولوژیستهای کمتری نیاز داشته و رادیولوژیستهای باقی مانده در بیمارستانها نیز به آموزش مجدد نیاز دارند. جفری هینتون (Geoffrey Hinton)، پدر علم یادگیری عمیق، معتقد است که (با توجه به پیشرفتهای احتمالی در ۵ یا ۱۰ سال آینده) بیمارستانها باید تعلیم رادیولوژیستهای جدید را کنار گذارند، زیرا وقت گیر و هزینه بر بوده و به زودی منسوخ خواهدشد.[63][64] یک اپراتور در JAMA، استدلال میکند که رشته پاتولوژی و رادیولوژی باید باهم ادغام شده و «متخصص اطلاعات» نام گیرد؛ و رادیولوژیستها باید با کامپیوتر جایگزین شوند. متخصصان اطلاعات باید منطق Bayesian، آمار و علوم داده، ژنومیک و زیست سنجی را بیاموزند. بازشناسی الگوی دستی برای رادیولوژیستها از اهمیت کمتری نسبت به قبل برخوردار است.[65]
جستارهای وابسته
منابع
- "Computer-aided Diagnosis: The Tipping Point for Digital Pathology". Digital Pathology Association. 27 April 2017.
- Bird, R. E.; Wallace, T. W.; Yankaskas, B. C. (1992). "Analysis of cancers missed at screening mammography". Radiology. 184: 613–617. doi:10.1148/radiology.184.3.1509041.
- Baker, J. A.; Rosen, E. L.; Lo, J. Y.; et al. (2003). "Computer-Aided Detection (CAD) in Screening Mammography: Sensitivity of Commercial CAD Systems for Detecting Architectural Distortion". American Journal of Roentgenology. 181: 1083–1088. doi:10.2214/ajr.181.4.1811083.
- Jang, H. J.; Lee, K. S.; Kwon, O. J.; et al. (1996). "Bronchioloalveolar carcinoma: focal area of ground-glass attenuation at thin-section CT as an early sign". Radiology. 199: 485–488. doi:10.1148/radiology.199.2.8668800.
- Suzuki, K.; Li, F.; Sone, S.; Doi, K. (2005). "Computer-aided diagnostic scheme for distinction between benign and malignant nodules in thoracic low-dose CT by use of massive training artificial neural network". IEEE Transactions on Medical Imaging. 24: 1138–1150. doi:10.1109/tmi.2005.852048.
- Lostumbo, A.; Suzuki, K.; Dachman, A. H. (2010). "Flat lesions in CT colonography". Abdom Imaging. 35: 578–583. doi:10.1007/s00261-009-9562-3.
- Echegaray, Sebastian; Gevaert, Olivier; Shah, Rajesh; Kamaya, Aya; Louie, John; Kothary, Nishita; Napel, Sandy (18 November 2015). "Core samples for radiomics features that are insensitive to tumor segmentation: method and pilot study using CT images of hepatocellular carcinoma". Journal of Medical Imaging. 2 (4): 041011. doi:10.1117/1.JMI.2.4.041011. PMC 4650964.
- Murphy, K.; van Ginneken, B.; Schilham, A. M.; et al. (2009). "A large-scale evaluation of automatic pulmonary nodule detection in chest CT using local image features and k-nearest-neighbour classification". Medical Image Analysis. 13: 757–770. doi:10.1016/j.media.2009.07.001.
- Suzuki, K.; Armato, 3rd, S. G.; Li, F.; Sone, S.; Doi, K. (2003). "Massive training artificial neural network (MTANN) for reduction of false positives in computerized detection of lung nodules in low-dose computed tomography". Med Phys. 30: 1602–1617. doi:10.1118/1.1580485.
- Chan, H. P.; Lo, S. C.; Sahiner, B.; et al. (1995). "Computer-aided detection of mammographic microcalcifications: pattern recognition with an artificial neural network". Med Phys. 22: 1555–1567. doi:10.1118/1.597428.
- Chen, S.; Suzuki, K.; MacMahon, H. (2011). "Development and evaluation of a computer-aided diagnostic scheme for lung nodule detection in chest radiographs by means of two-stage nodule enhancement with support vector classification". Med Phys. 38: 1844–1858. doi:10.1118/1.3561504. PMC 3069992. PMID 21626918.
- Papadopoulos, A.; Fotiadis, D. I.; Likas, A. (2005). "Characterization of clustered microcalcifications in digitized mammograms using neural networks and support vector machines". Artif Intell Med. 34: 141–150. doi:10.1016/j.artmed.2004.10.001.
- Wollenweber T.; Janke B.; Teichmann A.; Freund M. (2007). "Korrelation zwischen histologischem Befund und einem Computer-assistierten Detektionssystem (CAD) für die Mammografie". Geburtsh Frauenheilk. 67: 135–141. doi:10.1055/s-2006-955983.
- Taylor P, Potts HW (2008). "Computer aids and human second reading as interventions in screening mammography: Two systematic reviews to compare effects on cancer detection and recall rate". European Journal of Cancer. 44: 798–807. doi:10.1016/j.ejca.2008.02.016.
- Abe, Yoshiyuki; Hanai, Kouzo; Nakano, Makiko; Ohkubo, Yasuyuki; Hasizume, Toshinori; Kakizaki, Toru; Nakamura, Masato; Niki, Noboru; Eguchi, Kenji (2005-01-01). "A Computer-aided Diagnosis (CAD) System in Lung Cancer Screening with Computed Tomography". Anticancer Research. 25 (1B): 483–488. ISSN 0250-7005. PMID 15816616.
- Wu N, Gamsu G, Czum J, Held B, Thakur R, Nicola G (Mar 2006). "Detection of small pulmonary nodules using direct digital radiography and picture archiving and communication systems". J Thorac Imaging. 21 (1): 27–31. doi:10.1097/01.rti.0000203638.28511.9b. PMID 16538152.
- Giger, Maryellen Lissak; Doi, Kunio; MacMahon, Heber (1988-03-01). "Image feature analysis and computer-aided diagnosis in digital radiography. 3. Automated detection of nodules in peripheral lung fields". Medical Physics. 15 (2): 158–166. doi:10.1118/1.596247. ISSN 2473-4209.
- Ginneken, B. Van; Romeny, B. M. Ter Haar; Viergever, M. A. (2001-12-01). "Computer-aided diagnosis in chest radiography: a survey". IEEE Transactions on Medical Imaging. 20 (12): 1228–1241. doi:10.1109/42.974918. ISSN 0278-0062.
- Coppini, G.; Diciotti, S.; Falchini, M.; Villari, N.; Valli, G. (2003-12-01). "Neural networks for computer-aided diagnosis: detection of lung nodules in chest radiograms". IEEE Transactions on Information Technology in Biomedicine. 7 (4): 344–357. doi:10.1109/TITB.2003.821313. ISSN 1089-7771.
- Giger, M. L.; Bae, K. T.; MacMahon, H. (1994-04-01). "Computerized detection of pulmonary nodules in computed tomography images". Investigative Radiology. 29 (4): 459–465. doi:10.1097/00004424-199404000-00013. ISSN 0020-9996. PMID 8034453.
- Kanazawa, K.; Kawata, Y.; Niki, N.; Satoh, H.; Ohmatsu, H.; Kakinuma, R.; Kaneko, M.; Moriyama, N.; Eguchi, K. (1998-03-01). "Computer-aided diagnosis for pulmonary nodules based on helical CT images". Computerized Medical Imaging and Graphics (به English). 22 (2): 157–167. doi:10.1016/S0895-6111(98)00017-2. ISSN 0895-6111. PMID 9719856.
- Chen, Sheng; Zhong, Sikai; Yao, Liping; Shang, Yanfeng; Suzuki, Kenji (2016). "Enhancement of chest radiographs obtained in the intensive care unit through bone suppression and consistent processing". Physics in Medicine and Biology. 61 (6): 2283–2301. doi:10.1088/0031-9155/61/6/2283.
- Chen, S.; Suzuki, K. (2014-02-01). "Separation of Bones From Chest Radiographs by Means of Anatomically Specific Multiple Massive-Training ANNs Combined With Total Variation Minimization Smoothing". IEEE Transactions on Medical Imaging. 33 (2): 246–257. doi:10.1109/TMI.2013.2284016. ISSN 0278-0062.
- Suzuki, K.; Abe, H.; MacMahon, H.; Doi, K. (2006-04-01). "Image-processing technique for suppressing ribs in chest radiographs by means of massive training artificial neural network (MTANN)". IEEE Transactions on Medical Imaging. 25 (4): 406–416. doi:10.1109/TMI.2006.871549. ISSN 0278-0062.
- LOOG, M; VANGINNEKEN, B; SCHILHAM, A (2006-12-01). "Filter learning: Application to suppression of bony structures from chest radiographs". Medical Image Analysis (به English). 10 (6): 826–840. doi:10.1016/j.media.2006.06.002. ISSN 1361-8415.
- Chen, S.; Suzuki, K. (2013-02-01). "Computerized Detection of Lung Nodules by Means of #x201C;Virtual Dual-Energy #x201D; Radiography". IEEE Transactions on Biomedical Engineering. 60 (2): 369–378. doi:10.1109/TBME.2012.2226583. ISSN 0018-9294. PMC 4283823. PMID 23193306.
- doi:10.1016/j.crad.2018.02.009
- Suzuki, Kenji; Yoshida, Hiroyuki; Näppi, Janne; Dachman, Abraham H. (2006-10-01). "Massive-training artificial neural network (MTANN) for reduction of false positives in computer-aided detection of polyps: Suppression of rectal tubes". Medical Physics. 33 (10): 3814–3824. doi:10.1118/1.2349839. ISSN 2473-4209.
- Chaplot, S. ، LM Patnaik، و NR Jagannathan, طبقهبندی تصاویر مغز تشدید مغز با استفاده از موجک به عنوان ورودی برای پشتیبانی از دستگاه بردار و شبکه عصبی. پردازش و کنترل سیگنالهای بیولوژیکی، 2006 (1): ص. 86-92.
- Maitra, M. و A. Chatterjee، سیستم هوشمند مبتنی بر Slantlet برای طبقهبندی مغز تشدید مغناطیسی. پردازش و کنترل سیگنالهای بیولوژیکی، 2006 (1) (4): ص. 299-306.
- Wang, S.; Wu, W. (2010). "A Novel Method for Magnetic Resonance Brain Image Classification based on Adaptive Chaotic PSO". Progress in Electromagnetics Research. 109: 325–343.
- Zhang, Yudong; Wu, L. (2011). "Magnetic Resonance Brain Image Classification by an Improved Artificial Bee Colony Algorithm". Progress in Electromagnetics Research. 2011: 65–79.
- Saritha, M. ، KP Joseph، و AT Mathew, طبقهبندی تصاویر مغز مغناطیسی با استفاده از توطئههای عنکبوت مبتنی بر آنتروپی موجولی ترکیبی و شبکه عصبی احتمالی. نامههای شناخت الگو، 2013 (34) (16): ص. 2151-2156.
- Zhang, Yudong; Dong, Zhengchao; Ji, Genlin (2015). "Effect of spider-web-plot in MR brain image classification". Pattern Recognition Letters. 62: 14–16. doi:10.1016/j.patrec.2015.04.016.
- Zhang, Y.; Wang, S.; Ji, G.; Dong, Z. (2013). "Genetic Pattern Search and its Application to Brain Image Classification". Mathematical Problems in Engineering. 2013: 1–8. doi:10.1155/2013/580876.
- Das S.; Chowdhury M.; Kundu M.K. (2013). "Brain MR Image Classification Using Multiscale Geometric Analysis of Ripplet". Progress in Electromagnetics Research-Pier. 137: 1–17. doi:10.2528/pier13010105.
- Zhang, Y.; Wang, S. (2013). "An MR Brain Images Classifier System via Particle Swarm Optimization and Kernel Support Vector Machine". The Scientific World Journal. 2013: 9. doi:10.1155/2013/130134.
- Kalbkhani H.; Shayesteh M.G.; Zali-Vargahan B. (2013). "Robust algorithm for brain magnetic resonance image (MRI) classification based on GARCH variances series". Biomedical Signal Processing and Control. 8 (6): 909–919. doi:10.1016/j.bspc.2013.09.001.
- El-Dahshan E.S.A.; Mohsen H.M.; Revett K.; et al. (2014). "Computer-aided diagnosis of human brain tumor through MRI: A survey and a new algorithm". Expert Systems with Applications. 41 (11): 5526–5545. doi:10.1016/j.eswa.2014.01.021.
- Zhou, Xing-Xing (2015). "Detection of Pathological Brain in MRI Scanning Based on Wavelet-Entropy and Naive Bayes Classifier". Bioinformatics and Biomedical Engineering: 201–209. doi:10.1007/978-3-319-16483-0_20.
- Padma, A. و R. Sukanesh, جداسازی و طبقهبندی تصاویر سی تی مغز با استفاده از ترکیبی از ویژگیهای بافت آمار است. مجله علمی و مهندسی عربی، 2014. 39 (2): ص. 767-776.
- Zhang, Yudong; Wang, Shuihua; Dong, Zhengchao (2014). "Classification of Alzheimer Disease Based on Structural Magnetic Resonance Imaging by Kernel Support Vector Machine Decision Tree". Progress in Electromagnetics Research - Pier. 144: 185–191.
- Friston, K.; Poline, J-P.; Holmes, C.J. ,; Frith, C.D.; Frackowiak, R.S. (1996). "A multivariate Analysis of PET activation studies". Hum. Brain Mapp. 4: 140–151. doi:10.1002/(SICI)1097-0193(1996)4:2<140::AID-HBM5>3.0.CO;2-3.
- Martínez-Murcia, F.J.; Górriz, J.M.; Ramírez, J.; Puntonet, C.G.; Illán, I.A. (2013). "Functional activity maps based on significance measures and Independent Component Analysis". Computer Methods and Programs in Biomedicine. 111 (1): 255–268. doi:10.1016/j.cmpb.2013.03.015.
- Dong, Z.C. (2015). "Detection of subjects and brain regions related to Alzheimer's disease using 3D MRI scans based on eigenbrain and machine learning". Frontiers in Computational Neuroscience. 66 (9): 1–15. doi:10.3389/fncom.2015.00066.
- Zhang, J.; Yu, C.; Jiang, G.; Liu, W.; Tong, L. (2012). "3d texture analysis on mri images of alzheimer's disease". Brain imaging and behavior. 6 (1): 61–69. doi:10.1007/s11682-011-9142-3.
- Chupin, Marie; Gérardin, Emilie; Cuingnet, Rémi; Boutet, Claire; Lemieux, Louis; Lehéricy, Stéphane; Benali, Habib; Garnero, Line; Colliot, Olivier (2009). "Fully automatic hippocampus segmentation and classification in Alzheimer's disease and mild cognitive impairment applied on data from ADNI". Hippocampus. 19 (6): 579–587. doi:10.1002/hipo.20626.
- Martinez-Murcia, F.J.; Gorriz, J.M.; Ramirez, J.; Ortiz, A. (2016). "A Spherical Brain Mapping of MR Images for the Detection of Alzheimer's Disease". Current Alzheimer Research. 13 (5): 575–588. doi:10.2174/1567205013666160314145158. PMID 26971941.
- "EXINI Diagnostics".
- Huang, Kao and Chen (18 June 2007). "A Set of Image Processing Algorithms for Computer-Aided Diagnosis in Nuclear Medicine Whole Body Bone Scan Images". IEEE Transactions on Nuclear Science. 54: 514–522.
- Kaur, M; Talwar, R (2014). "Review on: blood vessel extraction and eye retinopathy detection" (PDF). International Journal of Computer Science and Information Technologies. 5 (6): 7513–7516.
- Tufail, A; Rudisill, C; Egan, C; Kapetanakis, VV; Salas-Vega, S; Owen, CG; Lee, A; Louw, V; Anderson, J (n.d.). "Automated diabetic retinopathy image assessment software: diagnostic accuracy and cost-effectiveness compared to human graders". Ophthalmology. 124: 343–351. doi:10.1016/j.ophtha.2016.11.014.
- Ahmad, A.; Mansoor, A. B.; Mumtaz, R.; Khan, M.; Mirza, S. H. (2014-12-01). "Image processing and classification in diabetic retinopathy: A review". 2014 5th European Workshop on Visual Information Processing (EUVIP): 1–6. doi:10.1109/EUVIP.2014.7018362.
- Fraz, M. M.; Barman, S. A.; Remagnino, P.; Hoppe, A.; Basit, A.; Uyyanonvara, B.; Rudnicka, A. R.; Owen, C. G. (2012-11-01). "An Approach to Localize the Retinal Blood Vessels Using Bit Planes and Centerline Detection". Comput. Methods Prog. Biomed. 108 (2): 600–616. doi:10.1016/j.cmpb.2011.08.009. ISSN 0169-2607.
- Priya, R; Aruna, P (2011). "Review of automated diagnosis of diabetic retinopathy using the support vector machine". International Journal of Applied Engineering Research, Dindigul. 1 (4): 844–862.
- Biradar, S; Jadhav, AS (2015). "A survey on blood vessel segmentation and optic disc segmentation of retinal images" (PDF). International Journal of Advanced Research in Computer and Communication Engineering. 4 (5): 21–26.
- Saleh, Marwan D.; Eswaran, C. (2012-10-01). "An Automated Decision-support System for Non-proliferative Diabetic Retinopathy Disease Based on MAs and HAs Detection". Comput. Methods Prog. Biomed. 108 (1): 186–196. doi:10.1016/j.cmpb.2012.03.004. ISSN 0169-2607.
- Antal, B.; Hajdu, A. (2012-06-01). "An Ensemble-Based System for Microaneurysm Detection and Diabetic Retinopathy Grading". IEEE Transactions on Biomedical Engineering. 59 (6): 1720–1726. arXiv:1410.8577. doi:10.1109/TBME.2012.2193126. ISSN 0018-9294.
- Patwari, Manjiri; Manza, Ramesh; Rajput, Yogesh; Saswade, Manoj; Deshpande, Neha (2013-10-01). "Review on Detection and Classification of Diabetic Retinopathy Lesions Using Image Processing Techniques". ResearchGate. 2 (10).
- Administrator (2015-05-20). "Review on: Detection of Diabetic Retinopathy using SVM and MDA". International Journal of Computer Applications. 117 (20).
- Espona, L.; Carreira, M. J.; Ortega, M.; Penedo, M. G. (2007-06-06). Martí, Joan, ed. Pattern Recognition and Image Analysis. Lecture Notes in Computer Science. Springer Berlin Heidelberg. pp. 178–185. doi:10.1007/978-3-540-72849-8_23#page-1. ISBN 978-3-540-72848-1.
- Paiva, Omir Antunes; Prevedello, Luciano M. (October 2017). "The potential impact of artificial intelligence in radiology". Radiologia Brasileira. 50 (5): V–VI. doi:10.1590/0100-3984.2017.50.5e1.
- Mukherjee, Siddhartha (27 March 2017). "A.I. Versus M.D." The New Yorker. Retrieved 3 February 2018.
- "Why scan-reading artificial intelligence is bad news for radiologists". The Economist. 29 November 2017. Retrieved 3 February 2018.
- Jha, Saurabh; Topol, Eric J. (13 December 2016). "Adapting to Artificial Intelligence". JAMA. 316 (22): 2353. doi:10.1001/jama.2016.17438.
