مدلسازی سیستمهای زیستی
مدلسازی سیستمهای بیولوژیکی از کاربردهای مهم زیستشناسی سیستمی و زیستشناسی ریاضیاتی است. هدف سیستمهای محاسباتی زیستی[1] پیادهسازی و استفاده از الگوریتمهای کارآمد، ساختمان دادهها، و ابزارهای تصویرسازی و ابزارهای ارتباطات با هدف شبیهسازی کامپبوتری سیستمهای زیستی است. این مدلسازی شامل استفاده از شبیهسازیهای کامپیوتری سیستمهای بیولوژیکی من جمله زیرسیستمهای سلولی (مثل شبکههای متابولیتها و آنزیمهای دخیل در متابولیسم (سوخت و ساز) گذرگاههای سیگنالینگ و شبکههای تنظیمی ژن) به منظور تحلیل و هم چنین تصویرسازی ارتبطات پیچیدهٔ بین این فرآیندهای سلولی است.
حیات مصنوعی یا تکامل مجازی در تلاش است که فرآیندهای تکاملی را به وسیلهٔ شبیهسازی کامپیوتری شکلهای سادهٔ (مصنوعی) زندگی درک کند.
بررسی اجمالی
همانطور که میدانیم ویژگیهای بروزیافته در یک سیستم پیچیده نتیجهٔ فعل و انفعال بین اجزای یکپارچه و سادهتر است. سیستمهای زیستی نمونههای مهم و متعددی از ویژگیهای ناشی از فعل و انفعالات پیچیده اجزا را آشکار میکنند. مطالعهٔ سنتی سیستمهای بیولوژیکی نیازمند روشهایی تقلیلی است که در آن مقادیر دادهها به صورت دستهبندی شده جمعآوری میشوند مثل غلظت در طول زمان در پاسخ به محرکی خاص.
کامپیوترها برای تحلیل و مدلسازی این دادهها حیاتی هستند. هدفساخت مدلهای بی درنگ و دقیقی از پاسخ یک سیستم به محرکهای محیطی و درونی است، مثل مدل سلول سرطانی به منظور یافتن ضعفها در گذرگاههای سیگنالینگ آن یا مدلسازی جهشهای کانال یونی برای مشاهدهٔ اثرات آن بر قلب و عملکرد قلب تپنده.
استانداردها
فرمت استانداردی که تاکنون به صورت گسترده برای ذخیرهسازی وتبادل مدلها پذیرفته شده، زبان نشانه گذاری زیست سیستمی (SBML)[2] است. وب سایت SBML.org شامل یک راهنما برای بسیاری از بستههای نرمافزاری مهم مورد استفاده در زیستشناسی سیستمی محاسباتی است. زبانهای دیگر نشانه گذاری با تاکیدات مختلف شامل BioPAX و CellML هستند.
وظایف خاص
مدل سلولی
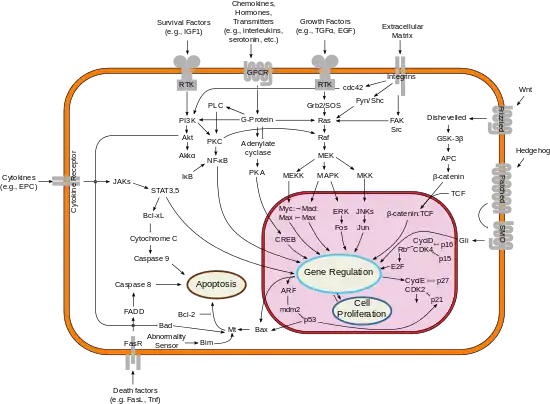
مدلسازی سلول یک وظیفهٔ چالشی زیستشناسی سیستمی و زیستشناسی ریاضیاتی است که شامل استفاده از شبیهسازیهای کامپیوتری تعداد زیادی از زیرسیستمهای سلولی مثل شبکههای متابولیتی و آنزیمهای دخیل در سوخت و ساز، مسیرهای سیگنالینگ و شبکههای تنظیم کنندهٔ ژن به منظور تحلیل و تصویرسازی ارتباطات پیچیدهٔ این فرآیندهای سلولی است.
شبکه پیچیدهای از واکنشهای بیوشیمیایی/فرآیندهای حمل و نقل و سازماندهی مکانی آنها، توسعه مدل پیشبینی کننده یک سلول زنده را همانطور که توسط بنیاد ملی علوم (NSF) در سال ۲۰۰۶ ذکر شده، تبدیل به یک چالش بزرگ برای قرن ۲۱ کردهاست.[3]
مدلی محاسباتی که برای سلول باکتری مایکوپلاسما ژنیتالیوم توسط دانشمندان دانشگاه استنفورد و مؤسسهٔ جی کریگ ونتر پیشنهاد شدهاست شامل تمام ۵۲۵ ژن آن، محصولات ژنی و تعاملات آنها است و در ۲۰ ژوئیه ۲۰۱۲ در مجلهٔ سلول چاپ شدهاست.[4]
یک مدل کامپیوتری پویا از سیگنالینگ درون سلولی، پایه و اساس داروهای Merrimack به منظور هدفیابی برای داروهای سرطان MM-111[5] بود.
محاسبات غشایی به منظور مدلسازی غشای سلول هستند
شبیهسازی موجودات چند سلولی
شبیهساز متن باز C. elegans در سطح سلولی توسط انجمن OpenWorm دنبال شدهاست. تا کنون موتور فیزیک Gepetto ساخته شده و مدلهای کانکتومهای عصبی و سلولهای عضلانی در فرمت NeuroML ایجاد شدهاست.[6]
تاشوندگی پروتئین
پیش بینی ساختار پروتئین در حقیقت پیشبینی ساختار سه بعدی یک پروتئین از توالی آمینو اسیدی یا پیشبینی ساختار سوم پروتئین از ساختار اولیه آن است. این یکی از مهمترین اهداف دنبال شده توسط بیوانفورماتیک و شیمی نظری است. پیشبینی ساختار پروتئین از اهمیت بالایی در پزشکی (برای مثال، در طراحی دارو) و بیوتکنولوژی (برای مثال، در طراحی آنزیمهای جدید) برخوردار است. هر دو سال، عملکرد روشهای فعلی در آزمایش CASP ارزیابی میشود.
مدل مغز
پروژه مغز آبی تلاش برای ایجاد یک مغز مصنوعی توسط مهندسی معکوس مغز پستانداران در سطح مولکولی است. هدف از این پروژه که در ماه مه سال ۲۰۰۵ توسط مؤسسه مغز و ذهن پلی تکنیک اکول لوزان، سوئیس، تأسیس شدهاست، مطالعه اصول معماری و کاربردی مغز است. این پروژه توسط مدیر مؤسسه، هنری مارکرام رهبری میشود. این شبیهسازی به کمک استفاده از یک ابرکامپیوتر ژن آبی و اجرای نرمافزار نورون مایکل هاینز انجام میشود که تنها شامل شبکههای عصبی مصنوعی ساده نیست بلکه شامل یک مدل بیولوژوکی نسبتاً واقعی از سلولهای عصبی است.[7][8] طرفداران این پروژه امیدوار هستند که توسط آن از ماهیت آگاهی مطلع شوند.. پروژه مغز بشر بر مبنای کار پروژه مغز آبی ساخته شدهاست.[9][10] این یکی از شش طرح آزمایشی است که در برنامه پژوهشی فن آوریهای ظهورشونده کمیسیون اروپا،[11] برای بودجه میلیارد یورویی رقابت میکند.
مدلسازی سیستم ایمنی بدن
دهه گذشته شاهد ظهور تعداد فزایندهای از شبیهسازیهای سیستم ایمنی بدن بود.[12][13]
کبد مجازی
پروژه کبد مجازی یک پروژهٔ ۴۳ میلیون یورویی است که بودجهٔ آن توسط دولت آلمان تأمین شده و از هفتاد گروه تحقیقاتی در سراسر آلمان تشکیل شدهاست. هدف از این پروژه ساخت مدل ریاضیاتی پویایی است که نشان دهندهٔ فیزیولوژی، مورفولوژی و عملکرد کبد انسان است.[14]
مدل درختی
درختان الکترونیکی (e-درختان) معمولاً با استفاده از سیستم های-ال رشد را شبیهسازی میکنند. سیستمهای ال در زمینه علم پیچیدگی و حیات بسیار مهم هستند. هنوز نیاز به ابداع یک سیستم پذیرفته شده جهانی برای توصیف تغییر در مورفولوژی گیاهی در سطح سلولی یا مدولار هستیم.[15] الگوریتمهای تولید درختی که بهطور گسترده پیادهسازی شدهاند در مقالات "Creation and Rendering of Realistic Trees", and Real-Time Tree Rendering توصیف شدهاند
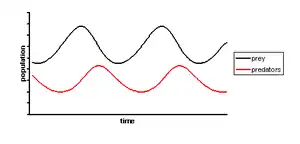
مدلهای اکولوژیکی
مدلهای اکوسیستم بیان ریاضی اکوسیستمها هستند. بهطور معمول آنها شبکههای غذایی پیچیده را به اجزای اصلی آنها یا سطوح غذایی ساده کرده و آن را به عنوان کمیت تعداد ارگانیسمها، زیست توده یا موجودی/غلظت برخی عناصر شیمیایی مربوطه (برای مثال کربن یا گونههای مغذی مانند نیتروژن یا فسفر) در نظر میگیرند.
مدلها در بوم آلاینده شناسی
ورود هر نوع ماده خارجی به آب، هوا و خاک به میزانی که کیفیت فیزیکی، شیمیایی یا بیولوژیکی اجزای محیط را تغییر داده و به حال موجودات زنده مضر باشد، آلودگی نام دارد. مباحثی مرتبط که در بوم آلاینده شناسی مطرح میشود شامل مواد آلاینده طبیعی، مواد آلاینده مصنوعی، تقسیمبندی آلودگیها، اثرات مواد آلاینده و آلودگی توسط مواد رادیواکتیو میباشد. هدف از مدلها در بوم آلاینده شناسی است فهم، شبیهسازی و پیشبینی اثرات ناشی از سموم در محیط زیست است. رایجترین مدلهای موجود اثرات را در یکی از سطوح متعدد و مختلف سازمانهای زیستی (به عنوان مثال موجودات زنده یا جمعیت) توصیف میکنند. توسعهٔ این مدلها در جهت پیشبینی اثرات در مقیاسهای زیستی از چالشهای موجود است. بوم آلایندهشناسی تعدادی از این مدلها را مورد بحث قرار داده و ارتباط به تعدادی دیگر را فراهم میکند.
مدلسازی بیماریهای عفونی
ظهور مجدد برخی از بیماریهای عفونی گذشته و پیدایش بیماریهای عفونی جدید توجه ویژه سامانههای بهداشتی را به مسایل مربوط به بیماریهای عفونی معطوف ساخته است این امکان وجود دارد که پیشرفت بسیاری از بیماریهای عفونی را برای اطلاع از احتمالهمه گیری یا برای کمک به مدیریت آنها توسط واکسیناسیون به صورت ریاضی مدل کرد. این زمینه در تلاش برای پیدا کردن پارامترهای مختلف بیماریهای عفونی و استفاده از این پارامترها برای محاسبه اثرات تودهای برنامهٔ واکسیناسیون است.
جستارهای وابسته
- تجسم دادههای زیستی
- شبیهسازی زیستی
- الگوریتم گیلیسپی
- نرمافزار مدلسازی مولکولی
- شبیهسازی تصادفی
منابع
- Andres Kriete, Roland Eils, Computational Systems Biology, Elsevier Academic Press, 2006.
- Klipp, Liebermeister, Helbig, Kowald and Schaber. (2007).
- American Association for the Advancement of Science
- Karr, J. (2012) A Whole-Cell Computational Model Predicts Phenotype from Genotype Cell
- McDonagh, CF (2012) Antitumor Activity of a Novel Bispecific Antibody That Targets the ErbB2/ErbB3 Oncogenic Unit and Inhibits Heregulin-Induced Activation of ErbB3.
- OpenWorm Downloads
- Graham-Rowe, Duncan.
- Palmer, Jason.
- «The Human Brain Project». بایگانیشده از اصلی در ۸ مه ۲۰۱۱. دریافتشده در ۳۰ دسامبر ۲۰۱۶.
- Video of Henry Markram presenting The Human Brain Project on 22 June 2012.
- FET Flagships Initiative homepage.
- Multi-criterion Evolutionary Algorithm with Model of the Immune System to Handle Constraints for Task Assignments - Springer
- "Computer Simulation Captures Immune Response To Flu". Retrieved 2009-08-19.
- «Virtual Liver Network». بایگانیشده از اصلی در ۳۰ سپتامبر ۲۰۱۲. دریافتشده در ۳۰ دسامبر ۲۰۱۶.
- "Simulating plant growth". Archived from the original on 9 December 2009. Retrieved 2009-10-18.
- Antmann, S. S.; Marsden, J. E.; Sirovich, L., eds. (2009). Mathematical Physiology (2nd ed.). New York, New York: Springer. ISBN 978-0-387-75846-6.
- Barnes, D.J.; Chu, D. (2010), Introduction to Modelling for Biosciences, Springer Verlag
- آشنایی با مدلساز بیماریهای عفونی توسط امیلیا Vynnycky و ریچارد G سفید. کتابی مقدماتی در رابطه با مدلسازی بیماریهای عفونی و کاربردهای آن.